김빛내리 서울대 교수 등 연구진, RNA-단백질 상호작용 원리 규명
 왼쪽부터 김빛내리 RNA 연구단장(공동교신저자), 배종우 연구원(제1저자), 김종서 연구위원(공동교신저자). 뒷쪽은 이번 연구에 사용한 질량분석기(Orbitrap Fusion Lumos, Thermo Fisher Scientific)다. 사진제공=기초과학연구원(IBS)
왼쪽부터 김빛내리 RNA 연구단장(공동교신저자), 배종우 연구원(제1저자), 김종서 연구위원(공동교신저자). 뒷쪽은 이번 연구에 사용한 질량분석기(Orbitrap Fusion Lumos, Thermo Fisher Scientific)다. 사진제공=기초과학연구원(IBS)
[아시아경제 김봉수 기자] 지난해 세계 최초로 코로나19 바이러스의 고해상도 유전자 지도를 제작해 주목을 받은 김빛내리 서울대 교수 등 한국 연구진들이 이번엔 생명현상 조절에 필수적인 RNA-단백질 상호작용의 비밀을 풀 열쇠를 찾아냈다.
기초과학연구원(IBS)은 김종서 RNA 연구단 연구위원(서울대 조교수)·김빛내리 단장(서울대 석좌교수) 연구팀이 세포 내 단백질에서 RNA와 결합을 형성하는 ‘RNA 결합자리’를 염기 종류에 따라 정확하게 찾아내는 기법을 개발했다고 25일 밝혔다. 생명현상 조절에 필수적인 RNA-단백질 상호작용의 원리 규명에 크게 기여할 것으로 기대된다.
RNA 결합자리의 동정은 세포 기능 조절을 규명하기 위한 핵심 단계라고 할 수 있다. RNA에 단백질이 결합하여 번역 효율, 안정성, 세포 내 위치 등 단백질 생산과정을 조절해 정상적인 유전자를 발현시키기 때문이다. 이때 RNA 결합자리를 확인하려면 작은 단백질 조각의 질량을 측정해, 해당 조각의 아미노산 및 단백질 내 위치를 추론하는 ‘질량분석’ 방법이 필요하다.
연구진은 지난해 짧은 파장의 자외선(UVC)과 불산을 적용, RNA 내 유라실과 단백질의 교차결합을 형성하여 RNA 결합자리를 찾는 기술을 개발했다. 그런데 이 기술로는 RNA-단백질 상호작용의 복합적 파악이 어려웠다. RNA-단백질 교차결합 형성 효율이 매우 낮으며, 가능한 RNA 염기 종류도 유라실 하나뿐이었기 때문이다.
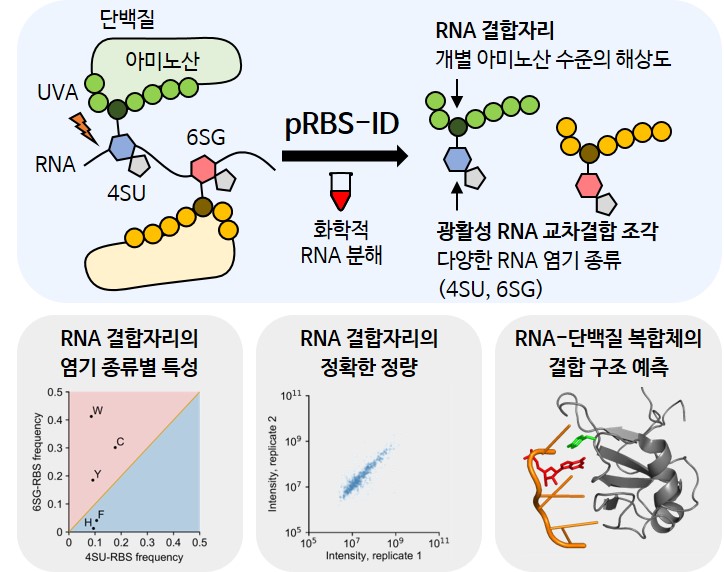 광활성 RNA를 이용한 pRBS-ID 연구 결과 모식도.
pRBS-ID를 위해서는 먼저 UVA를 이용해 광활성 RNA-단백질 간 공유결합을 형성한다. 그 뒤 단백질은 펩타이드로 만들고, RNA는 불산을 이용해 화학적으로 분해한다. 이후 질량분석을 수행하면 펩타이드에 교차결합한 광활성 RNA 조각의 질량으로부터 RNA 결합자리를 개별 아미노산 수준의 고해상도로 동정할 수 있다. 연구진은 pRBS-ID를 활용한 본 연구에서 RNA 결합자리의 염기 종류별 특성을 파악했고, RNA 결합자리를 정량값을 정확히 측정했으며, RNA-단백질 복합체의 구조 데이터에 기반한 RNA-단백질 상호작용 분석의 기반을 마련했다.
광활성 RNA를 이용한 pRBS-ID 연구 결과 모식도.
pRBS-ID를 위해서는 먼저 UVA를 이용해 광활성 RNA-단백질 간 공유결합을 형성한다. 그 뒤 단백질은 펩타이드로 만들고, RNA는 불산을 이용해 화학적으로 분해한다. 이후 질량분석을 수행하면 펩타이드에 교차결합한 광활성 RNA 조각의 질량으로부터 RNA 결합자리를 개별 아미노산 수준의 고해상도로 동정할 수 있다. 연구진은 pRBS-ID를 활용한 본 연구에서 RNA 결합자리의 염기 종류별 특성을 파악했고, RNA 결합자리를 정량값을 정확히 측정했으며, RNA-단백질 복합체의 구조 데이터에 기반한 RNA-단백질 상호작용 분석의 기반을 마련했다.
연구진은 긴 파장의 자외선(UVA)과 광활성 RNA 표지법을 활용해 작년 연구의 한계를 극복했다. 우선 세포 내 RNA를 4-티오유리딘과 6-티오구아노신 두 종류의 광활성 염기로 표지했다. 이후 UVA를 이용해 결합된 단백질과 특이적 교차결합을 유도하고, 단백질에 결합된 긴 RNA를 불산 처리하여 화학적으로 가수 분해했다. 그리고 질량분석 방법으로 남은 RNA 조각의 분자량을 확인했다. 이 정보를 토대로 4-티오유리딘과 6-티오구아노신의 각 염기 종류에 해당하는 특이적 RNA 결합자리를 고효율로 동정해냈다. 또한, 데이터 분석 프로그램을 발전시켜 RNA 결합자리를 포함하는 펩타이드를 정확히 정량했다.
이로써 연구진은 염기 종류 및 교차결합 방식에 따른 RNA-단백질 상호작용 양상의 차이를 면밀히 규명했다. 이는 새로 발견한 광활성 RNA 결합자리 뿐 아니라, 기존 UVC를 이용한 RNA 결합자리까지 망라하는 성과다. 나아가 RNA-단백질 복합체 구조 데이터를 접목해, 질량분석으로 규명한 RNA 결합자리가 밝혀진 단백질-RNA 구조와 일치하는 염기 종류와 상호작용함을 확인했다. 또 단백질의 구조가 불안정하여 기존 데이터로 파악할 수 없었던 RNA-단백질 상호작용도 RNA 결합자리를 통해 밝혀냈다.
연구팀은 "이번에 개발한 질량분석 기법과 새로 동정한 3000개 이상의 RNA 결합자리 정보는 RNA-단백질 상호작용에 의한 생명현상 조절 이해에 활용될 수 있다"며 "특히 광활성 RNA의 대사표지와 최근 획기적으로 개선된 단백질 구조 예측 프로그램들을 접목하면, RNA-단백질 상호작용의 동적 변화와 분자적 기전을 세밀히 이해할 수 있을 것"이라고 설명했다.
지금 뜨는 뉴스
이번 연구 결과는 국제학술지 네이처 커뮤니케이션스(Nature Communucations)(IF 14.92)에 지난 15일 온라인 게재됐다.
김봉수 기자 bskim@asiae.co.kr
<ⓒ투자가를 위한 경제콘텐츠 플랫폼, 아시아경제(www.asiae.co.kr) 무단전재 배포금지>







![전문가 4인이 말하는 '의료 생태계의 대전환'[비대면진료의 미래⑥]](https://cwcontent.asiae.co.kr/asiaresize/319/2026013014211022823_1769750471.png)